主な違い サンガー配列決定とパイロシークエンシングの違いは、サンガー配列決定はジデオキシ チェーン ターミネーション法を使用する DNA 配列決定アプローチであるのに対し、パイロシークエンスは合成による配列決定の原則に基づく DNA 配列決定アプローチです。 したがって、サンガー シーケンスでは、ヌクレオチドの識別は全 DNA フラグメントの増幅後にキャピラリー電気泳動によって行われますが、パイロシーケンスでは、合成中にピロリン酸を放出してヌクレオチドの識別が行われます。
サンガー配列決定とパイロ配列決定は、DNA 配列決定の 2 つの方法です。前者はほとんどのターゲットの「ゴールド スタンダード」であり、後者は従来のサンガー シーケンス法に代わる最初の方法です。
対象となる主な分野
1. サンガー シーケンスとは
– 定義、プロセス、重要性
2. パイロシーケンシングとは
– 定義、プロセス、重要性
3. サンガー シーケンスとパイロシーケンスの類似点は何ですか
– 共通機能の概要
4. サンガーシーケンシングとパイロシーケンシングの違いは何ですか
– 主な相違点の比較
主な用語
DNA シークエンシング、PCR、ピロリン酸、ピロシークエンシング、サンガー シークエンシング、感度
サンガー シーケンスとは
サンガー配列決定は、1977 年に Fredric Sanger によって最初に開発された DNA 配列決定の第一世代の方法です。さらに、サンガー配列決定の基礎はジデオキシ チェーン ターミネーション法です。
サンガーシーケンシング – 手順
サンガー シーケンスでは、DNA ポリメラーゼが in vitro DNA 合成中に鎖末端ジデオキシヌクレオチド(ddNTP)を選択的に取り込みます。したがって、ジデオキシヌクレオチド (ddNTPs) は PCR によってアンプリコンに蛍光標識されます。ここでは、ddATP は緑色の色素で標識されています。 ddGTP は黄色の色素で標識されています。 ddCTP は青色で標識され、ddTTP は赤色の色素で標識されています) 次に、蛍光標識されたヌクレオチドを検出しながら、得られたアンプリコンをキャピラリー電気泳動で分離します。
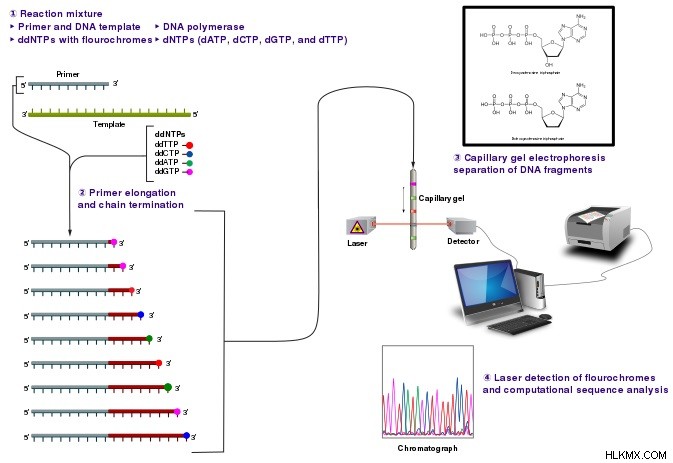
図 1:サンガー シーケンス法
サンガー配列決定 – 重要性
しかし、サンガー配列決定法には、より長い配列決定出力を処理できない、少数のサンプルの並列分析ができない、サンプル調製の完全な自動化ができない、コストが高い、配列決定が難しいなど、いくつかの制限があります。エラー、感度の低下 (10 ~ 20%)、低レベルの突然変異対立遺伝子の検出には不十分などです。これらの制限にもかかわらず、多くの臨床手順におけるシーケンスの「ゴールド スタンダード」です。
パイロシーケンシングとは
パイロシーケンシングは、従来のサンガー法に代わる最初の方法です。王立工科大学(KTH)で開発された次世代シーケンシングの一種です。さらに、この方法は、プライマー指向の DNA ポリメラーゼが触媒するヌクレオチド取り込み中に放出されるピロリン酸(PPi)のルミノメトリー検出に基づいています。
パイロシーケンシング – 手順
一般に、この方法では取り込まれたヌクレオチドを正確に検出するために 4 つの酵素が使用されます。それらは、DNAポリメラーゼ、ATPスルフリラーゼ、ルシフェラーゼ、およびアピラーゼです。さらに、シーケンシング プライマーは、ビオチン標識された一本鎖 DNA のテンプレートにハイブリダイズします。さらに、4 つのデオキシヌクレオチド三リン酸(dNTP)、アデノシン 5' ホスホ硫酸(APS)、およびルシフェリンが反応混合物中の基質です。
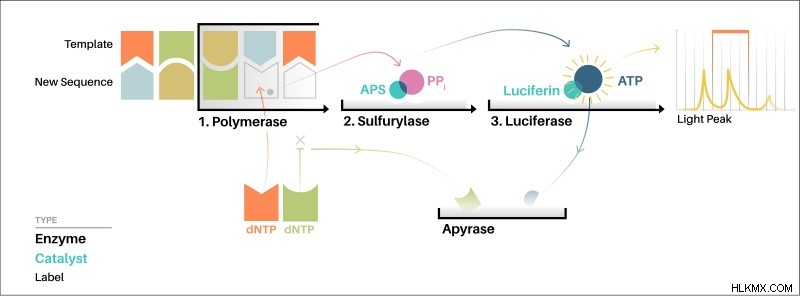
図 2:パイロシーケンシング法
重合カスケードが始まると、ポリメラーゼによるヌクレオチド取り込みの結果として無機PPiが放出されます。ただし、放出されたPPiの量は、各サイクルで取り込まれたヌクレオチドの量と等モルです。続いて、ATPスルフリラーゼは、放出されたPPiをAPSの存在下で定量的にATPに変換します。生成された ATP は、ルシフェラーゼ酵素によって媒介されるルシフェリンからオキシルシフェリンへの変換を促進します。また、この反応は、ATP の量に比例して可視光を生成します。次に、この光は 560 nm の波長で検出できます。
さらに、アピラーゼ酵素の主な機能は、反応混合物中の ATP および取り込まれていない dNTP を継続的に分解することです。したがって、新しい dNTP は、65 秒の特定の時間間隔で一度に 1 つずつ反応に追加する必要があります。追加されたヌクレオチドが既知であるため、テンプレートの配列を決定することができます。
パイロシーケンシング – 重要性
さらに、パイロシーケンシングは、高精度、並列処理、容易に自動化できる、広く適用可能な技術です。また、標識プライマー、標識ヌクレオチド、およびゲル電気泳動の使用を回避します。さらに、確認シーケンスと de novo シーケンスの両方に適しています。さらに、パイロシーケンシングの主な重要な特徴は、そのシーケンスの深さであり、これにより、高感度でバリアントを検出できます。ただし、この手法の主な欠点は、最大数百の塩基をシーケンスするのに適していることです。
サンガー シーケンスとパイロシーケンシングの類似点
- サンガー シーケンスとパイロシーケンスは、 DNA 配列決定。
- 彼らは、目的の DNA フラグメントのヌクレオチド配列。
- どちらも小さな DNA フラグメントのシーケンスに適しています.
- ただし、依存する独自のアプリケーションがあります。シーケンス手順と利点について。
サンガー シーケンスとパイロシーケンスの違い
定義
サンガー配列決定とは、鎖末端のジデオキシヌクレオチドを選択的に組み込むことによる DNA 配列決定の方法を指し、パイロシークエンスとは、合成による配列決定の原則に基づく DNA 配列決定の方法を指します。 .
配列の種類
サンガー配列決定は第一世代の配列決定アプローチであり、パイロシークエンスは第二世代の配列決定アプローチである次世代の配列決定化学です。
相関
さらに、サンガー シーケンスは従来の方法であり、ほとんどのターゲットの「ゴールド スタンダード」ですが、パイロシーケンスは従来のシーケンス方法に代わる最初の方法です。
発明
1977 年、フレデリック サンガーとその同僚は、1977 年にサンガー シーケンスを開発した最初の人物であり、一方、ポール ニレンと彼の学生であるモスタファ ロナギは、ストックホルムの王立工科大学でパイロシーケンスを開発した最初の人物でした。 1996.
商品化
サンガー シーケンスは Applied Biosystems によって最初に商品化されましたが、パイロシーケンスは Roche 454 および GS FLX Titanium プラットフォームで使用されています。
原則
何よりも、サンガー シーケンスとパイロシーケンスの主な違いは、サンガー シーケンスがジデオキシ チェーン ターミネーション法を使用するのに対して、パイロシーケンスは合成によるシーケンスの原則に基づいていることです。
ヌクレオチドの同定
サンガー シーケンスでは、ヌクレオチドの識別は DNA フラグメント全体の増幅後にキャピラリー電気泳動によって行われますが、パイロシーケンスでは、ヌクレオチドの識別はピロリン酸の放出によって行われます。合成。
検出
さらに、サンガー シーケンスには蛍光の検出が含まれますが、パイロシーケンスには 560 nm の可視光の検出が含まれます。
DNA 断片の長さ
さらに、サンガー シーケンスは最大 800 ~ 1000 塩基対を読み取ることができ、パイロシーケンスは最大 300 ~ 500 塩基対を読み取ることができます。
意義
サンガーシーケンシングは多くのステップを伴う複雑なプロセスですが、パイロシーケンシングはステップ数が少ない単純なプロセスです。
感度
また、サンガー法とパイロシーケンシングのもう 1 つの違いは、サンガー法は感度が低く、パイロシークエンス法は感度が高いことです。
結論
サンガー配列決定は、第一世代の配列決定アプローチであり、従来の配列決定方法です。また、これは多くのターゲットにとって「ゴールド スタンダード」です。ただし、キャピラリー電気泳動に続いてジデオキシ チェーン ターミネーション法を使用します。一方、パイロシーケンシングはサンガーシーケンシングに代わる最初のシーケンシングであり、次世代シーケンシングの一種です。さらに、感度が高く、カバーするステップが少なくなります。一般的には、DNA断片をそのまま合成する際に塩基を決定するSequencing by synthesis法が用いられます。したがって、サンガーシーケンシングとパイロシーケンシングの主な違いは、シーケンシングの方法とその利点です。