主な違い UPGMA と近隣の結合ツリーの間の重要な点は、UPGMA が a平均リンケージ法に基づく凝集型階層的クラスタリング法 一方、隣人結合 ツリーは、最小進化基準に基づく反復クラスタリング手法です。 さらに、UPGMA は根付きの系統樹を生成しますが、近隣結合ツリー法は根のない系統樹を生成します。 UPGMA メソッドは等しい進化率を前提としているため、枝の先端は等しくなりますが、隣接結合ツリー法では不均等な進化率が許容されるため、枝の長さは変化量に比例します。
UPGMA (算術平均による重み付けされていないペア グループ法) と隣接結合 (NJ) ツリーは、距離行列から系統樹を構築する 2 種類のアルゴリズムです。一般に、UPGMA は単純で高速ですが、信頼性の低い方法ですが、近隣結合ツリー法は比較的高速な方法であり、UPGMA 法よりも優れた結果が得られます。
対象となる主な分野
1. UPGMA とは
– 定義、方法、意義
2. Neighbor Joining Tree とは
– 定義、方法、意義
3. UPGMA と近隣結合ツリーの類似点は何ですか
– 共通機能の概要
4. UPGMA と近隣結合ツリーの違いは何ですか
– 主な相違点の比較
主な用語
凝集クラスタリング法、距離行列、近隣結合ツリー、系統樹 
UPGMAとは
UPGMA (算術平均による重み付けされていないペア グループ法) は、Sokal と Michener に起因する単純な凝集型の階層的クラスタリング手法です。これは、ルート化されたウルトラメトリック 系統樹を構築するための最も簡単で最速の方法です。ただし、この方法の主な欠点は、すべての系統で同じ進化速度を仮定していることです。これは、これらの系統の突然変異率が時間の経過とともに一定であることを意味します。これは「分子時計仮説」とも呼ばれています。さらに、同様の距離でツリー内のすべてのブランチを生成します。ただし、すべての系統で同じ突然変異率を持つことは難しいため、実際には、UPGMA 方式では信頼性の低いツリー トポロジが生成されることが多くなります。
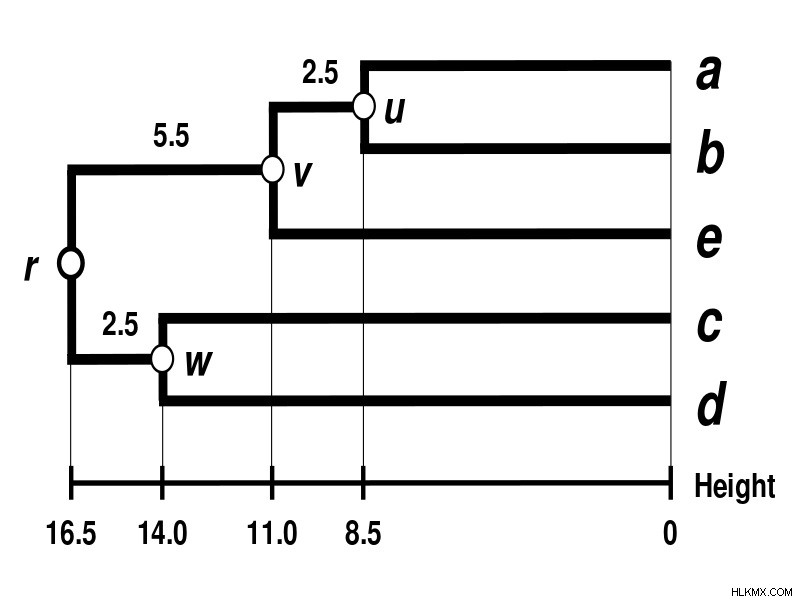
図 1:UPGMA メソッド
さらに、UPGMA メソッドはペアごとの距離のマトリックスから始まります。最初は、それぞれの種が独自のクラスターであると想定しています。次に、距離マトリックスの最小距離値を持つ最も近い 2 つのクラスターを結合します。さらに、平均を取ることによって関節ペアの距離を再計算します。次に、すべての種が 1 つのクラスターに接続されるまで、アルゴリズムはこのプロセスを繰り返します。
近隣結合ツリーとは
近隣結合 (NJ) ツリー法は、系統樹の構築に使用される最新の凝集クラスタリング法です。 1987 年に斉藤成也と根井正敏によって開発されました。ただし、ルートのない系統樹を構築します。さらに、超距離距離を必要とせず、星分解法を使用します。さらに、近隣結合ツリー アルゴリズムは、系統の進化速度の変動を調整します。したがって、それは未解決の星のようなツリーから始まります。
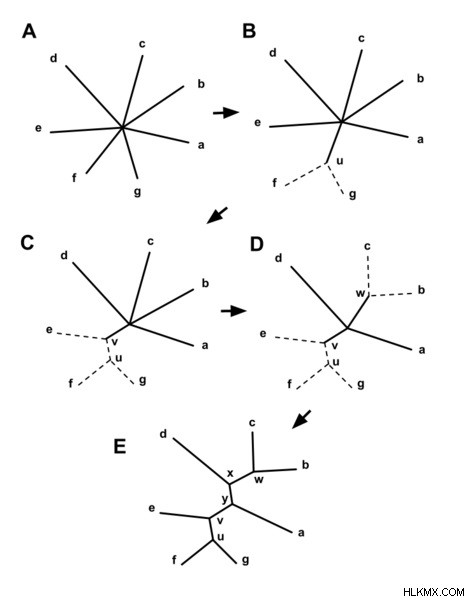
図 2:近隣結合ツリーの構築
さらに、隣接結合木法では、行列 Q は現在の距離に基づいて計算されます。次に、距離が最も短い系統のペアを選択して、新しく作成されたノードに結合します。ただし、このノードはセントラル ノードと接続されています。その後、アルゴリズムは各系統から新しいノードまでの距離を計算します。次に、各系統から新しいノードまでの外側からの距離を計算します。最後に、計算された距離に基づいて、結合された隣接ノードを新しいノードに置き換えます。
UPGMA と隣接ツリーの類似点
- UPGMA と隣接結合ツリーは、入力として距離行列を取り、系統樹を構築する 2 つのアルゴリズム。一般に、距離行列は 2D 行列です。これは、一連の点のペアごとの距離を含む配列です。
- 関連するタンパク質または DNA 配列は、距離行列を構築するための尺度として使用できます。
- どちらも凝集体です(ボトムアップ)クラスタリング メソッド。
- これらは、計算量が少ないより高速なメソッドです高い。
- したがって、それらは大規模に適用できますデータセット。
- さらに、次の場合、どちらの方法でもより良い結果が得られます他のタイプの入力を使用する方法と比較して。
- 単一のツリーを生成するように設計されていますが、 、場合によっては複数のトポロジが生成され、データ入力順序に基づいて「無秩序」な動作が発生します。
- ブートストラップ値は、ノード/クレード形成の確率を確認します。
UPGMA と Neighbor Joining Tree の違い
定義
UPGMA は、距離行列から根付きの系統樹を構築するための直接的なアプローチを指し、隣接結合木は、星を介して根を持たない系統樹を構築するための新しいアプローチを指します。木。
開発者
UPGMA メソッドは 1958 年に Sokal と Michener によって開発され、隣接結合木 は 1987 年に 斉藤成也と根井正敏によって開発されました。
意義
さらに、UPGMA は平均リンケージ法に基づく凝集型の階層的クラスタリング法であり、隣接結合ツリーは最小進化基準に基づく反復クラスタリング法です。
系統樹の種類
UPGMA 法は根のある系統樹を構築しますが、隣接結合木法は無根の系統樹を構築します。
距離の種類
さらに、UPGMA アルゴリズムでは距離がウルトラメトリックである必要がありますが、隣接木を結合するアルゴリズムでは距離が習慣的である必要があります。
系統樹の枝の性質
UPGMA メソッドは進化の速度が等しいと仮定しているため、枝の先端は等しくなります (ルートから先端までの枝の長さは同じです)。隣接結合ツリー法では不均等な進化率が許容されるため、枝の長さは変化量に比例します。
スピード
UPGMA はシンプルで高速な方法ですが、隣接結合ツリーは比較的高速な方法です。
信頼性
さらに、UPGMA は信頼性の低い方法ですが、近隣結合ツリーはより良い結果をもたらします。
結論
UPGMA は、進化距離データに基づいて系統樹を構築する 2 つのアルゴリズムのうちの 1 つです。さらに、同様の枝の長さを持つ根のある系統樹を構築します。さらに、距離行列から系統樹を構築するためのシンプルで高速、かつ最も信頼性の高いアルゴリズムです。一方、隣接結合木は、距離行列から系統樹を構築するために使用される 2 番目の方法です。ただし、枝の長さが進化中の変化量を反映する根のない系統樹が生成されます。また、このアルゴリズムは比較的速度が遅くなりますが、最も信頼性の高い系統樹を構築します。したがって、UPGMA と近隣結合ツリーの主な違いは、系統樹の機能とアルゴリズムの機能です。